Proteomic Pathology
Methodenforschung
Die Forschung in der Pathologie analysiert die zellulären Vorgänge, die zur Entstehung und Progression von Krankheiten führen. In diesem Zusammenhang werden Zellen und Gewebe zunehmend als ein kompliziertes Netzwerk verstanden, in dem eine Vielzahl biologischer Prozesse gleichzeitig ablaufen und gemeinsam das biologische Verhalten bestimmen. Proteine sind an allen zellulären Prozessen beteiligt und somit sind zum Verständnis der zellulären Funktionen das Verständnis der zeitlich und örtlich differentiellen Protein- Expression, der Protein-Lokalisation, der Protein-Interaktion, der posttranslationalen Protein-Modifikation und der Protein-Aktivität essentiell.
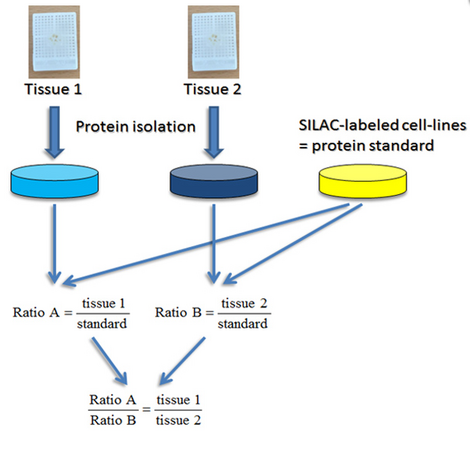
Quantitative Massenspektrometrie
Die globale Analyse der zellulaäen Proteinzusammensetzung würde die Sichtweite auf das zelluläre Netzwerk sehr erweitern, ist technisch jedoch anspruchsvoll. Ein großer Durchbruch in diese Richtung war die Einführung von Microarrays zur globalen Quantifizierung der mRNA-Expression. Obwohl dies zu einem starken Anstieg in unserem Wissen über viele zellulären Funktionen geführt hat, wäre die direkte Untersuchung der Proteinexpression häufig vorteilhaft, weil die mRNA- oft nicht gut mit der Proteinexpression korrelliert und posttranslationale Proteinmodifikationen wie Phosphorylierung mit Microarrays nicht erfasst werden können. Die Massenspektrometrie stellt eine Technologie dar, die es erlaubt, Proteine direkt, global und unvoreingenommen zu untersuchen.
Für die massenspektrometrischen Analysen von Gewebs- und Zelllysaten arbeiten wir in enger Kooperation und vielen gemeinsamen Projekten mit Dr. Christof Lenz und Prof. Dr. Henning Urlaub von der Proteomanalyse-Facility, Universitätsmedizin Göttingen und der Bioanalytischen Massenspektrometrie am Max-Planck-Institut für biophysikalische Chemie in Göttingen, wo die Analysen erfolgen, und mit Dr. Thomas Oellerich und Prof. Dr. Hubert Serve von der Medizinischen Klinik II, Universitätsklinikum Frankfurt zusammen.
Allgemeine Einführung in die proteomische quantitative Massenspektrometrie
Das zu untersuchende Proteingemisch wird zur massenspektrometrischen Analyse zunächst proteolytisch verdaut und die Peptide mittels Elektrospray-Ionisierung (ESI) ionisiert. In einem Orbitrap Massenspektrometer oszillieren die Peptide in einer bestimmten, von Masse und Ladung des Peptides abhängigen Frequenz um eine Ionenfalle. Dadurch können die Masse im Verhältnis zur Ladung (m/z) und die Intensität jedes Peptides bestimmt werden. In einem zweiten Messschritt wird die Sequenz des Peptides bestimmt, was als MS/MSAnalyse bezeichnet wird. Dazu werden die Peptide zunächst in eine Kollisionskammer überführt, wo sie fragmentiert werden. Durch die erneute Messung der Masse der Bruchstücke kann die Aminosäuresequenz des Peptides bestimmt werden. Dadurch ergibt sich der besondere Vorteil, dass a priori nicht bekannt sein muss, welche Proteine gemessen werden sollen, sondern das Massenspektrometer potentiell jedes beliebige Protein identifizieren kann.
Kinaseaktivität - PamGene

Die Aktivität eines Proteins wird von einer Vielzahl von Faktoren wie Expression, verschiedenen posttranslationalen Modifikationen oder Interaktion mit anderen Proteinen reguliert. Häufig stellt die abberante Aktivität von onkogen-wirksamen Kinasen eine Schlüsselrolle in der malignen Entartung einer Zelle dar. Ebenso ist es die Aktivität einer Kinase, die entscheidend ist für das Ansprechen auf einen spezifischen Kinase-Inhibitor als gezielte Krebs-Therapie.
Bei der Kinaseaktivitätsmessung mittels PamChips der Firma PamGene werden Peptide durch die Kinaseaktivität des zu untersuchenden Lysates phosphoryliert und mittels Fluoreszenzfarbstoff-markierter Antikörper sichtbar gemacht. Der Vorteil dabei ist, dass direkt die Aktivität der Kinasen im Lysat bestimmt wird. Ein weiterer Vorteil ist, dass Inhibitoren mit in das Lysat gegeben werden können, so dass direkt das Ansprechen auf einen spezifischen Kinaseinhibitor beobachtet werden kann.
Aktuelle Projekte
Aktuell werden in unserer Arbeitsgruppe mit den genannten Methoden verschiedener histologische und molekulare Subtypen des Lungenkrebs analysiert. Dabei arbeiten wir mit Dr. Alexander Emmert, Dr. Mark Hinterthaner, PD Dr. Bernd Danner und Prof. Dr. med. Dipl. Phys. Friedrich A. Schöndube von der Klinik für Thorax-, Herz- und Gefäßchirurgie zusammen. In Zukunft sollen die proteomischen Analysen auf weitere Malignom-Entitäten ausgedehnt werden.
Kooperationen
Proteomische Massenspektrometrie
Jasmin Corso
Prof. Dr. Henning Urlaub
Bioanalytische Massenspektrometrie Gruppe
Max Planck Institut für biophysikalische Chemie Göttingen
https://www.mpibpc.mpg.de/de/urlaub
Dr. Christof Lenz
Prof. Dr. Henning Urlaub
Proteomics Service Facility
Universitätsmedizin Göttingen
http://www.med.uni-goettingen.de/de/content/forschung/1998.html
Dr. med. Thomas Oellerich
Prof. Dr. med. Hubert Serve
Medizinischen Klinik II - Hämatologie und Onkologie
Universitätsklinikum Frankfurt
http://www.kgu.de/?id=3102
Funktionelle Bildgebung
Dr. Gertrude Bunt
Institut für Neuropathologie
Molecular and Optical Live Cell Imaging
Universitätsmedizin Göttingen
http://www.med.uni-goettingen.de/de/content/forschung/6656.html
Prof. Dr. Fred Wouters
Institut für Neuropathologie
Laboratory for Molecular and Cellular Systems
Universitätsmedizin Göttingen
http://www.uni-goettingen.de/de/58060.html
Thorax-Herz-Gefäßchirurgie
Dr. med. Alexander Emmert
Dr. med. Marc Hinterthaner
PD Dr. med. Bernd Danner
Prof. Dr. med. Dipl. Phys. Friedrich A. Schöndube
Klinik für Thorax-, Herz- und Gefäß-Chirurgie
Universitätsmedizin Göttingen
http://thg.med.uni-goettingen.de/index.php/de/
Kontakt
Kontaktinformationen
- Telefon: +49 551 3965051
- E-Mail-Adresse: hanibal.bohnenberger(at)med.uni-goettingen.de
Publikationen zum Thema
- Oellerich T, Oellerich MF, Engelke M, Münch S, Mohr S, Nimz M, Hsiao HH, Corso J, Zhang J, Bohnenberger H, Berg T, Rieger MA, Wienands J, Bug G, Brandts C, Urlaub H, Serve H. (2013) β2 integrin-derived signals induce cell survival and proliferation of AML blasts by activating a Syk/STAT signaling axis. Blood;121(19):3889-99, S1-66
- Lösing M, Goldbeck I, Manno B, Oellerich T, Schnyder T, Bohnenberger H, Stork B, Urlaub H, Batista FD, Wienands J, Engelke M. (2013) The Dok-3/Grb2 protein signal module attenuates Lyn kinase-dependent activation of Syk kinase in B cell antigen receptor microclusters. J Biol Chem;288(4):2303-13
- Oellerich T, Bremes V, Neumann K, Bohnenberger H, Dittmann K, Hsiao HH, Engelke M, Schnyder T, Batista FD, Urlaub H, Wienands J. (2011) The B-cell antigen receptor signals through a preformed transducer module of SLP65 and CIN85. EMBO J;30(17):3620-34
- Bohnenberger H, Oellerich T, Engelke M, Hsiao HH, Urlaub H, Wienands J. (2011) Complex phosphorylation dynamics control the composition of the Syk interactome in B cells. Eur J Immunol;41(6):1550-62
Das könnte Sie auch interessieren